DNA甲基化(5mC)是哺乳动物细胞中重要的表观遗传修饰,在基因表达调控和转座子沉默中发挥关键作用。长散在核元件(Long Interspersed Nuclear Element-1, LINE-1)是基因组中一类序列高度同源但甲基化修饰存在差异的重复元件。传统方法(如MeDIP-seq)仅能无差别涵盖全基因组的甲基化DNA,而基于dCas9的邻近标记技术(如dCas9耦联标记酶)虽能靶向特定DNA序列,却无法区分其甲基化状态。而如何同时识别这些元件的DNA序列和DNA的甲基化状态并捕获其结合的调控蛋白,一直是表观遗传学领域的挑战。

2025年4月18日,浙江大学基础医学院干细胞与再生医学系/良渚实验室/附属第一医院的钱鹏旭研究员联合基础医学院细胞生物学系陈宝惠研究员与中国医学科学院血液病医院王洪研究员团队,在Nature Communications期刊在线发表了题为Selective identification of epigenetic regulators at methylated genomic sites by SelectID的研究论文。该研究开发了一种新的邻近标记方法,命名为SelectID(Selective profiling of epigenetic control at genome targets identified by dCas9)。该方法通过将甲基化DNA结合结构域(MBD)整合至dCas9介导的邻近标记系统,实现在特定甲基化DNA区域的活细胞成像和调控蛋白捕获(图1,2),并成功鉴定出CHD4作为甲基化LINE-1(L1)转座子的抑制因子。该方法为表观遗传学中DNA甲基化修饰区域的调控机制解析提供新的思路。
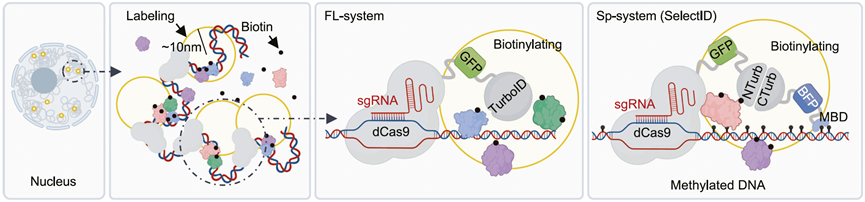
图1. SelectID技术通过dCas9和MBD双靶向体系实现甲基化区域的活细胞成和选择性蛋白标记的原理示意图
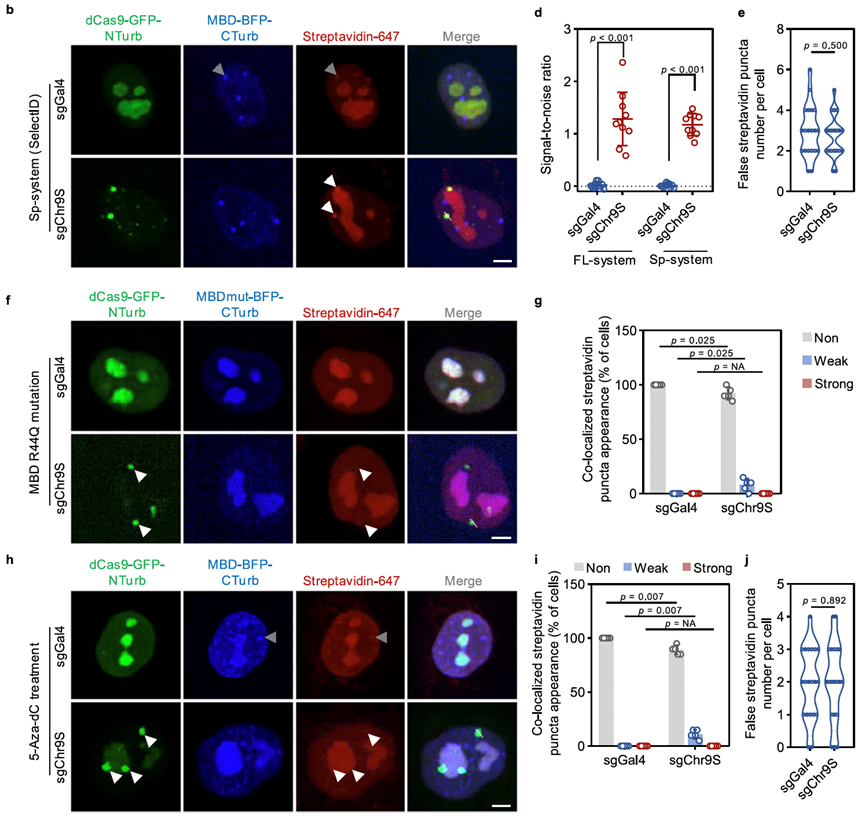
图2. SelectID实现对9号染色体卫星序列区域的活细胞标记和成像
LINE-1(L1)是人类基因组中唯一具有自主转座能力的逆转录转座子,约占基因组的20%,其异常激活可导致基因组不稳定和疾病发生。DNA甲基化(尤其是靠近5'端区域的5mC)是宿主抑制L1转座的重要机制,但此前缺乏识别特定甲基化位点结合蛋白的技术。研究团队将dCas9介导的Split-TurboID系统与甲基化DNA结合结构域(MBD)耦联,同时靶向年轻的L1元件(dCas9-GFP-NTurb)和特异性结合甲基化CpG位点(MBD-BFP-CTurb),且仅在dCas9与MBD同时存在时激活邻近标记,实现了选择性的蛋白捕获。通过整合优化的微量质谱技术(micro-DIA),SelectID仅需百万数量级的细胞即可完成甲基化L1元件的蛋白质组分析,有效地降低了背景噪音。在L1元件的甲基化5'UTR区域,SelectID鉴定出染色质重塑因子CHD4是一个关键抑制蛋白。RNA-seq和ChIP-seq证实CHD4通过直接结合甲基化L1元件的5'UTR抑制其表达(图3)。
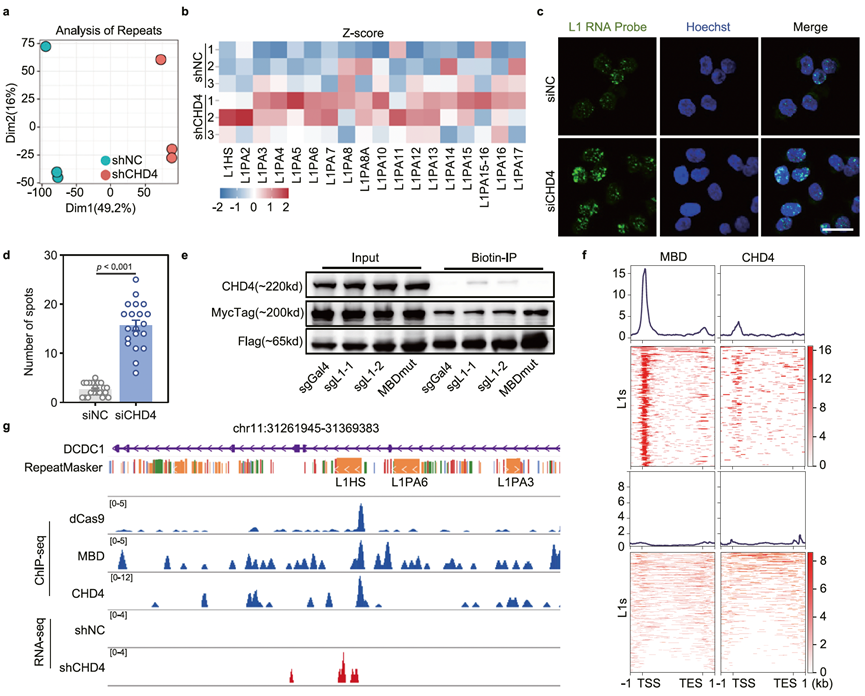
图3. CHD4通过结合甲基化L1的5'UTR抑制其转录活性
SelectID在活细胞中实现了DNA序列与甲基化状态的协同识别与标记,克服了传统方法无法区分表观修饰的局限。其应用可扩展至其他表观标记(如组蛋白修饰)的研究,为表观遗传学中染色质修饰区域的调控机制解析提供新的思路,但针对单一位点的精准捕获仍是当前技术难点。
浙江大学基础医学院博士后钱文畅、浙一附院特聘研究员姜蓬垒、浙江大学基础医学院博士生扶玉娟和中国医学科学院血液病医院的牛明明副研究员为该工作的第一作者。钱鹏旭研究员、陈宝惠研究员和中国医学科学院血液病医院的王洪研究员为该工作的通讯作者。本研究得到国家自然科学基金和中国博士后科学基金的资助,同时感谢浙江大学医学院公共技术平台和浙江大学附属第一医院中心实验室的技术支持。



