单细胞蛋白质组学已广泛应用于当前生物医药研究,但其数据处理长期面临方法选择的困境。一方面,处理流程众多且性能高度地依赖于所研究的具体数据集,增大了对处理流程的选择难度;另一方面,不当的处理易在下游分析中引入偏差,导致生物学解读失真以及研究结论不可重复。

10月16日,浙江大学药学院朱峰教授和付婷婷博士团队在Nature Protocols发表题为“navigating the data processing for cytometry-based single-cell proteomics”的研究论文,提出面向单细胞蛋白组学的数据处理工作流优选方法ANPELA(https://idrblab.org/anpela/)。该方法以机器学习与多维度评估为支撑,聚焦细胞亚群鉴定与伪时间轨迹推断两项关键任务,在提升处理稳健性与准确性的同时,为药理机制解析与精准医学研究提供可复用、可推广的单细胞蛋白组学数据处理技术路径。
保障高准确度的细胞亚群鉴定
研究人员开展了新冠病毒感染者与健康人群外周血细胞亚群鉴定研究,验证了此项工作所提出方法的效果。针对36例感染者与45名健康人的单细胞蛋白质组学数据,开展了大规模的工作流优选。结果显示,若不加选择地使用领域内常用的处理流程,细胞类型注释的误差可能超出预期(错误率超过20%)。相比之下,研究人员开发的ANPELA可将错误率大幅降低(下降幅度可达6% ~ 20%)。在对CD4⁺ T细胞、CD8⁺ T细胞与嗜碱性粒细胞等关键细胞亚群的鉴定中,ANPELA实现近乎完美的识别(错误率低于1%)。
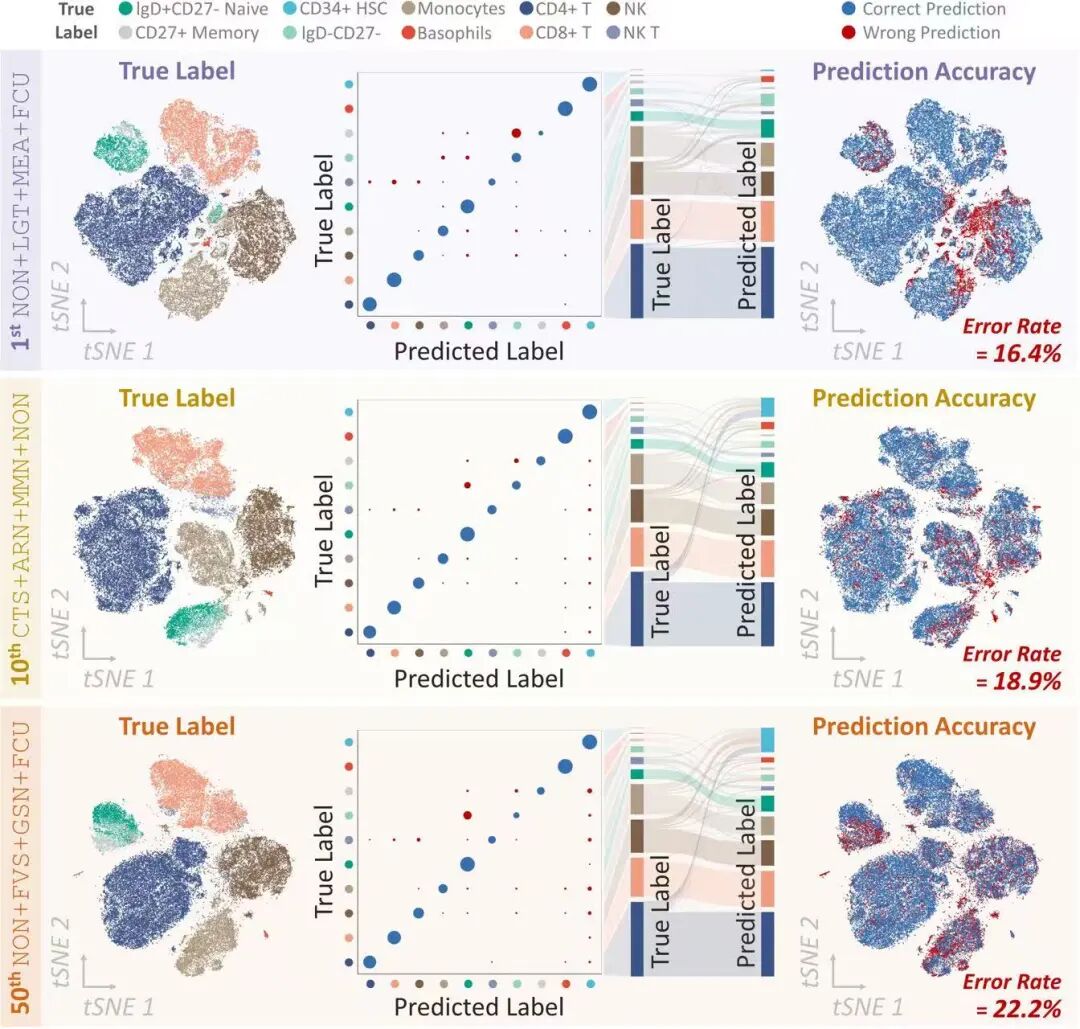
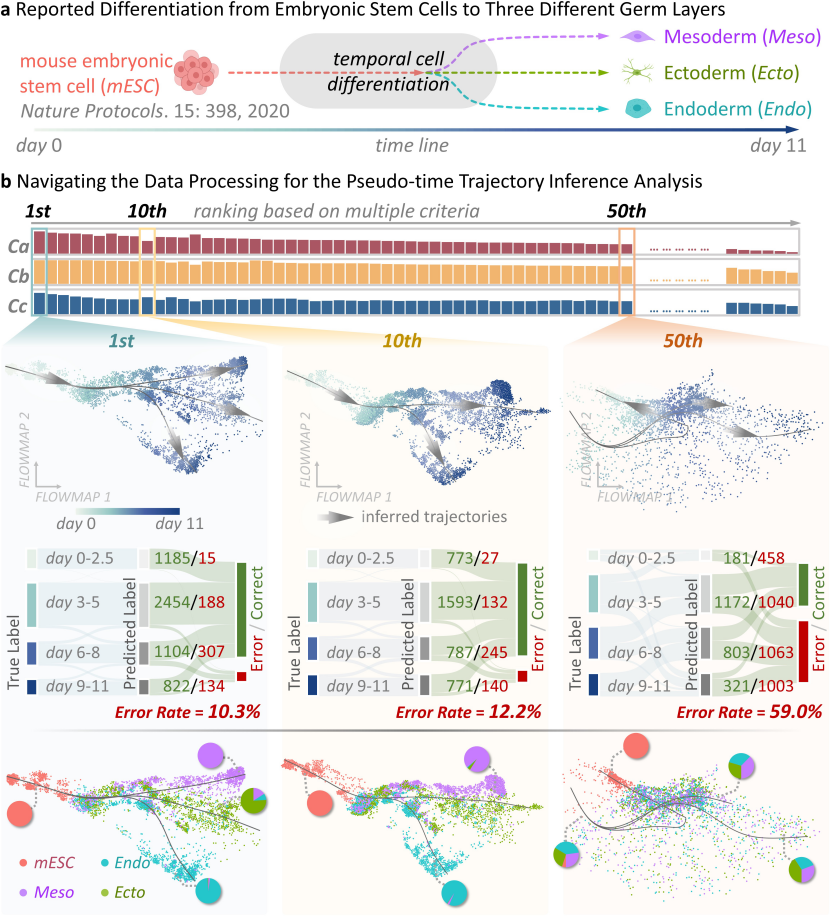
实现高可信度的细胞轨迹推断
在小鼠胚胎干细胞分化的单细胞蛋白组学数据上,研究团队进一步验证了所提出方法对伪时间轨迹推断的显著增益。该数据覆盖连续11天的多时间点采样,理想的轨迹应自胚胎干细胞出发,沿三条支路分别分化为外胚层、中胚层与内胚层,并在时间序列上与真实生物进程相一致。然而,若直接采用领域内常用的处理流程,分析结果会出现严重偏差,不仅轨迹的起点与终点错误,伪时间与真实时间的误差也超过50%。在部分伪时间与真实时间的误差较低的数据处理结果中,其对应的细胞分化轨迹亦频繁缺失中胚层分支。
相比之下,研究人员开发的ANPELA所推荐的最优流程能够稳定地重建与先验知识高度吻合的三分支发育轨迹,起点与终点分别对应小鼠胚胎干细胞和三类胚层细胞,并在定量评估中实现伪时间与真实时间仅相差10.3%,显著优于领域内常用流程,为疾病发生发展的解析提供了更为可信的细胞分化图景。
浙江大学药学院为本论文的第一署名单位,浙江大学博士生孙怀诚、周圆和本科生蒋若愚为共同第一作者,朱峰教授以及付婷婷博士为共同通讯作者。



